
现代健身文化鼓励人们追求多肌肉,少脂肪,追求轮廓分明的身体形态和线条,这意味着健康、强壮、力量、吸引力和人体美。

图1:力训研究所女成员
对于增肌的原理,大多数健身爱好者练了一辈子,增肌增了一辈子,都是稀里糊涂的,很多健身博主/科普者也是一知半解。
听他们讲增肌的原理,一会“适应性增粗”,一会“撕裂后超量恢复”,一会“肌肉没有办法被迫生长”,让人觉得捉急,因为这不包含任何信息量,跟没说一样。
要把原理讲清楚,那就非常复杂了。关键词:蛋白质合成、机械张力、细胞信号、DNA转录。
一、大多健身者对蛋白质存在误解大多说健身者都以为,我们吃下去的蛋白质作为原料,来构成我们的肌肉,这显然不对,因为这混淆了“蛋白质”和“氨基酸”。我们构筑身体,用的是氨基酸,不是蛋白质。
蛋白质由大量的氨基酸和键构成,是结构非常复杂的大分子物质[1]。我们举两个具有代表性的例子。第一个,人体血红蛋白(图2)。
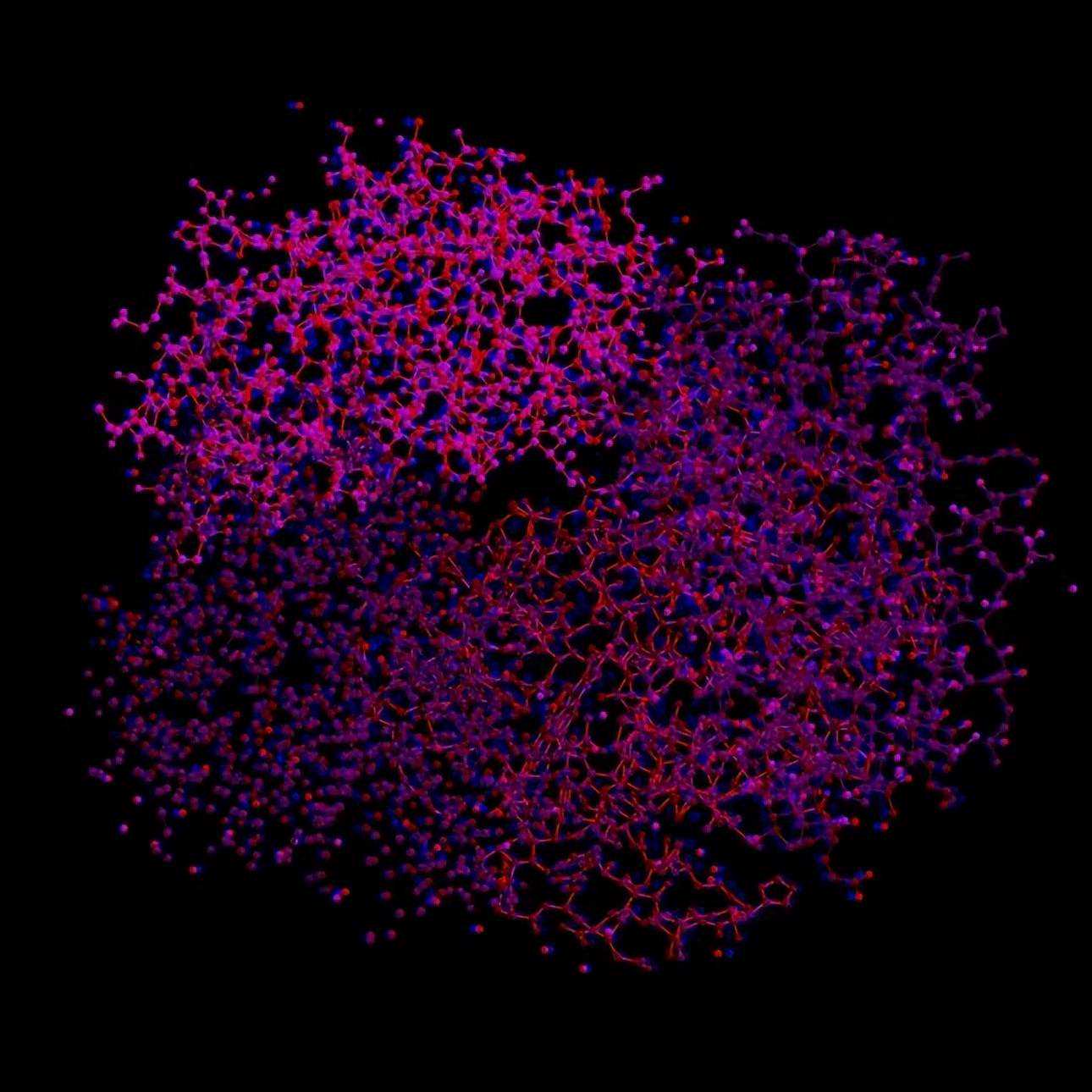
图2-1
第二个例子,某些真核生物体内的核糖体蛋白。
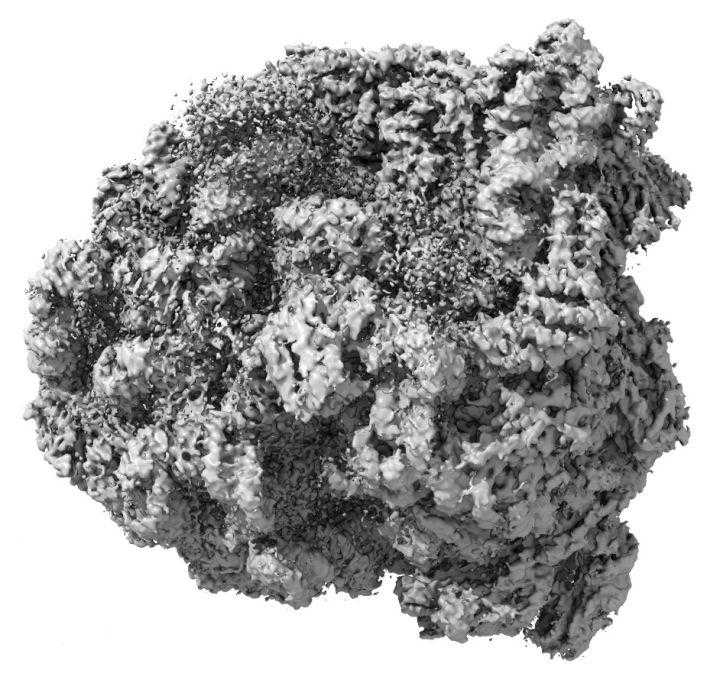
图2-2
图2-3
大家可以看出,蛋白质具有极其复杂的结构,我们根本不可能直接使用结构如此复杂的大分子物质来构建我们的身体,我们得先把蛋白质消化、拆分。
在消化和拆分的过程中,人利用各种胃蛋白酶,如胰蛋白酶[2][3][4][5]、肠蛋白酶[6][7][8][9]等,把食物中的蛋白质水解[10][11][12]成为最基本单位——氨基酸[13][14][15]、或多个氨基酸组成的短肽[16][17][18],然后才能用它们用来构筑我们的身体。
二、增肌,到底增是哪里?很多健身者都能回答:增的是肌纤维。
然后呢?没了。
大多数人都知道“增粗肌纤维”、增加肌肉中的蛋白质合成,但肌纤维是如何变粗的,蛋白质合成到底在肌纤维的哪里,却说不清楚。
对于我们来说,首先要搞清楚肌纤维的结构,肌纤维中的蛋白质增到了哪里。在一般人的认知中,细胞可能像个球一样,圆圆的,中间是细胞核。

图3:细胞
但肌细胞(肌细胞就是肌纤维)不是个球状的构成,而是像长长的管子一样。肌细胞表面是细胞膜,里面主要有更多更细的“管子”:肌原纤维,它外面包裹着肌浆网,细胞里还有线粒体、细胞核等等。
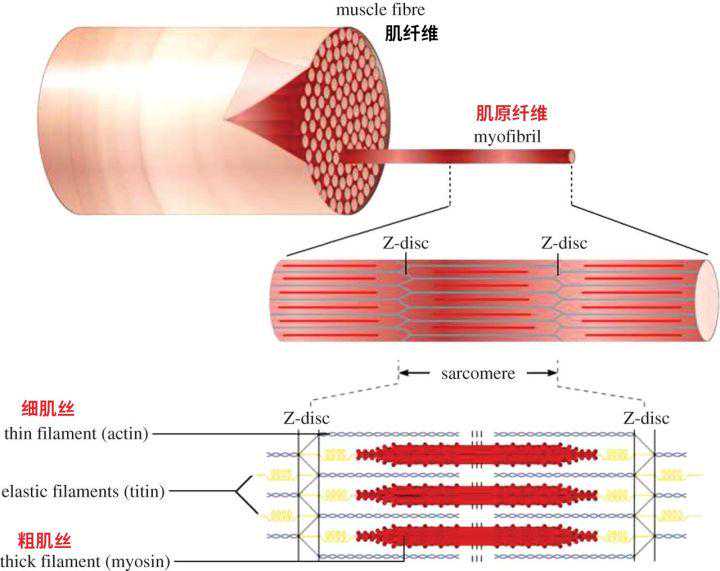
图4:肌肉结构
我们的肌肉之所以能够收缩,主要是因为肌纤维内的肌原纤维,它的内部含有更多更细的“肌丝”:粗肌丝、细肌丝[5,6]。
肌肉收缩时,在神经系统释放的生物电的刺激下[19][20][21][22],粗细肌丝之间的“锁”被打开[23][24][25][26][27],ATP氧化释放能量,带动粗/细肌丝相互“滑行”,肌肉缩短,完成收缩[28][29]。
肌动蛋白[30][31][32]构成我们肌纤维中的细肌丝,肌球蛋白[33][34][35][36]构成粗肌丝。例如人心肌中的肌球蛋白复合物:
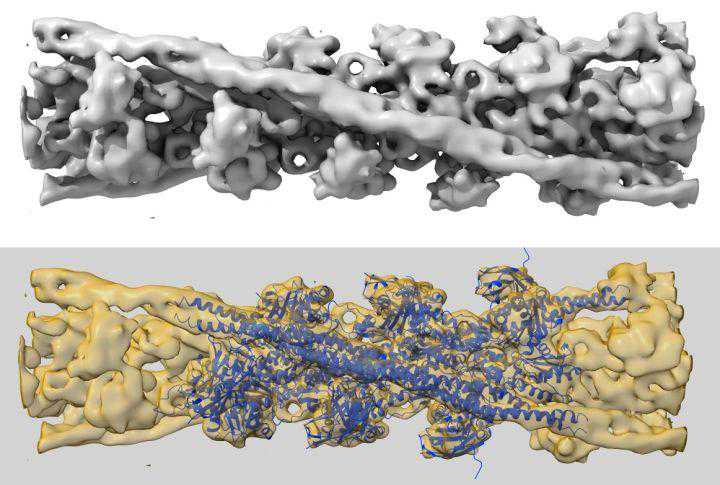
图5:人心肌中肌球蛋白复合物
现在大家应该明白了,所谓增肌,主要增的是肌原纤维上的粗、细肌丝上的蛋白。
三、肌原纤维内的蛋白质是怎么来的?许多人简单的认为,肌纤维中的蛋白质不就是吃下去的蛋白质分解成氨基酸组成的。这个说法没错,但几乎就是废话,因为氨基酸是如何组成蛋白质,这才是关键。
与大多数人想象的不同,实际上,蛋白质的种类非常多[37][38][39][40]。多到什么程度呢?真核生物一个细胞内的蛋白质,就多达几万种。
蛋白质的结构非常复杂[41][42][43],在空间中呈现立体的几何形态[44][45],具有多层的扭曲和折叠性状[46][47][48][49][50];只要稍微有一点变化,它的功能、特性和稳定性就可能发生变化[51][52][53]。

图6:胰岛素降解酶
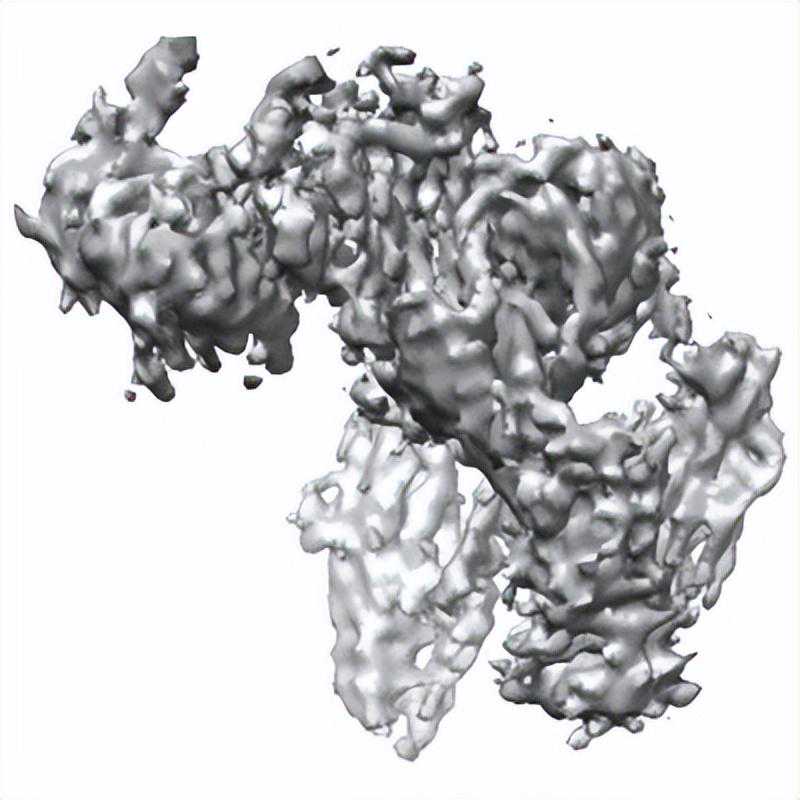
图7:IGF-1受体与胰岛素复合物

图8:亚硝酸盐还原酶
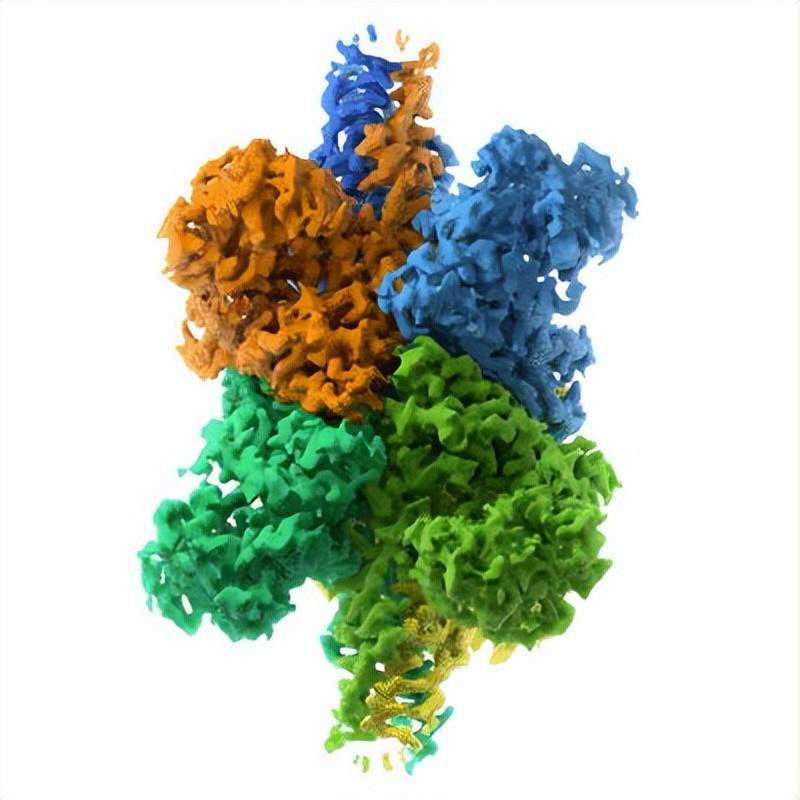
图9:谷氨酸脱氢酶
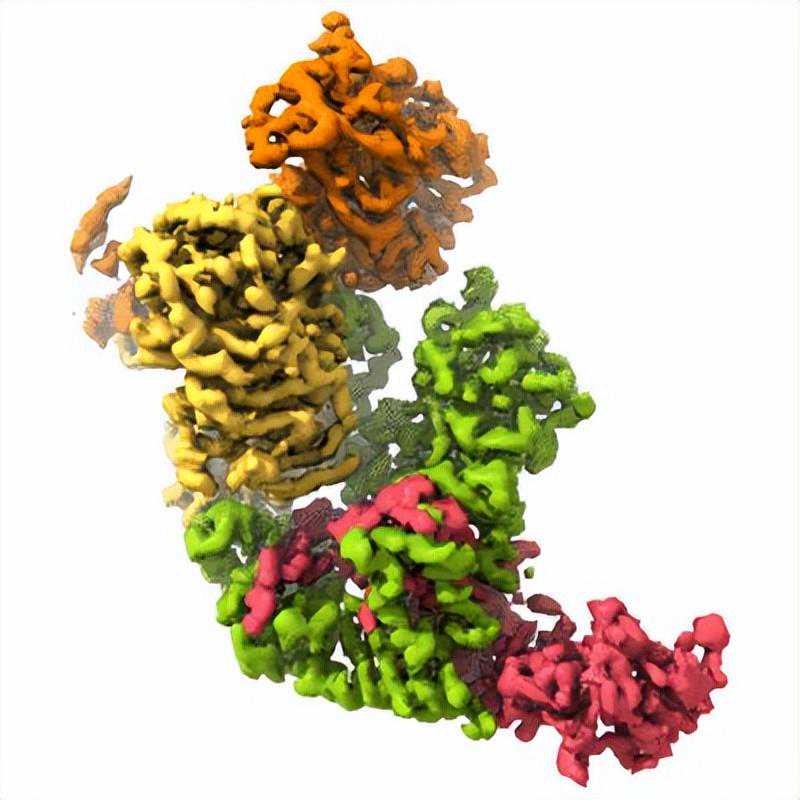
图10:E3连接酶泛素
蛋白质的结构非常复杂,当然也包括我们的肌肉。人肌肉里的蛋白质是大量氨基酸构成的生物大分子物质[54][55][56][57],人肌肉的肌球蛋白(及其结合物)在仪器的眼光下看上去长这样。
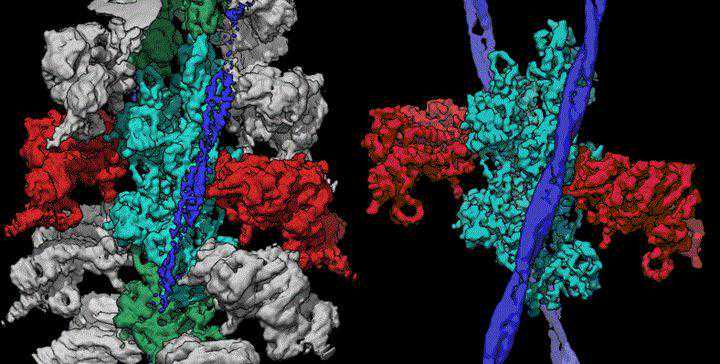
图11:人肌球蛋白复合物
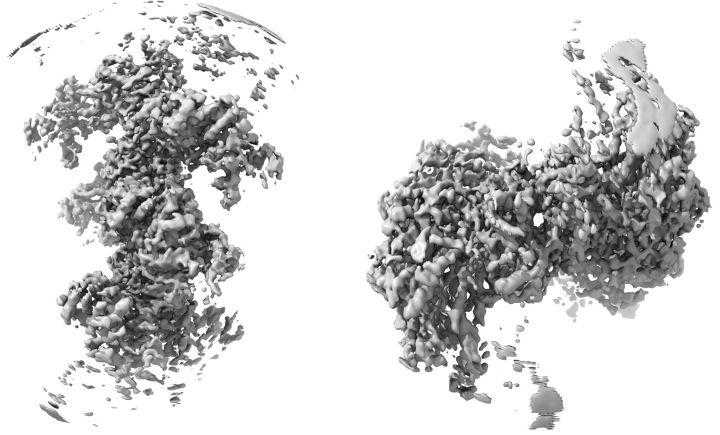
图12:人肌球蛋白复合物
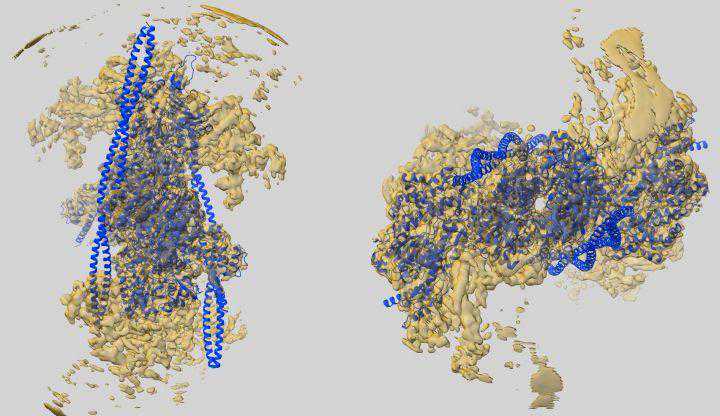
图13:人肌球蛋白复合物
由于人体肌肉中的蛋白质结构如此复杂,那么很显然,大量的氨基酸绝不可能凭白无故的、在没有指引的前提下,就能按照某种预先设置好的方式来构建如此复杂的大分子蛋白质。
这就像你有大量的砖石,但是用砖石制造建筑,需要设计图,并不是把砖石胡乱堆在一起就是建筑了。氨基酸组成蛋白质也是一样的道理。毫无疑问,有什么东西在引导它们。
答案是mRNA。
mRNA如同一条链子,上面预留了不同类型的氨基酸的结合区(密码子)。身体把大量的氨基酸运输过来,每个氨基酸可以对号入座,“组装”到这条链子上。
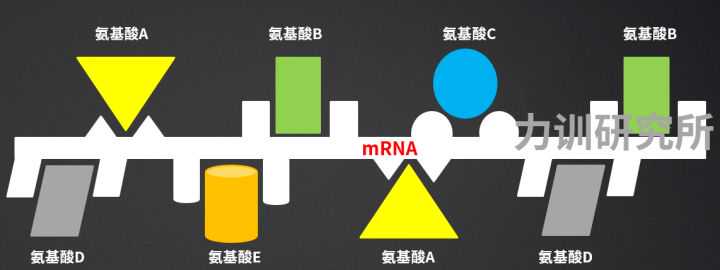
图14:mRNA
当然,光是组装还远远不够。组装好了以后,这只是形成了蛋白质的雏形而已,蛋白质有四级空间结构,从宏观上看,是多重折叠的。例如人体血红蛋白,3D看是这样:

图15:人体血红蛋白
但是,人体血红蛋白,如果用图形表示,在教科书上,是这样的:
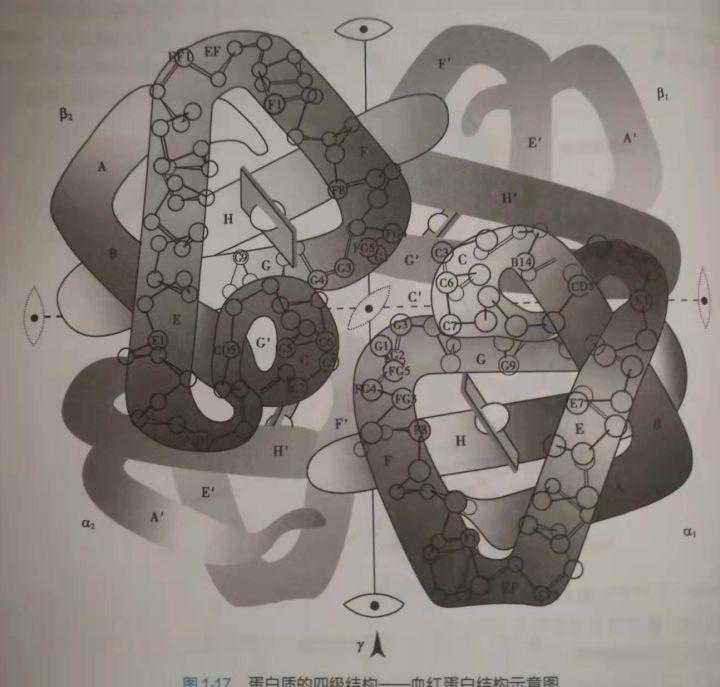
图16:人体血红蛋白
仅仅氨基酸“组装”到mRNA上还不够,还有折叠[58][59][60][61]、修饰[62][63][64][65]、转运[66][67]等许多工作要做;我们把mRNA变成蛋白质(的雏形)这一步,叫做翻译"[68][69][70][71][72]。
那mRNA哪来的呢?是DNA以它自身为模板,复制出来的(单螺旋结构)。这一部叫做转录[73][74][75][76];从DNA到蛋白质,宏观上主要是转录和翻译这两步。
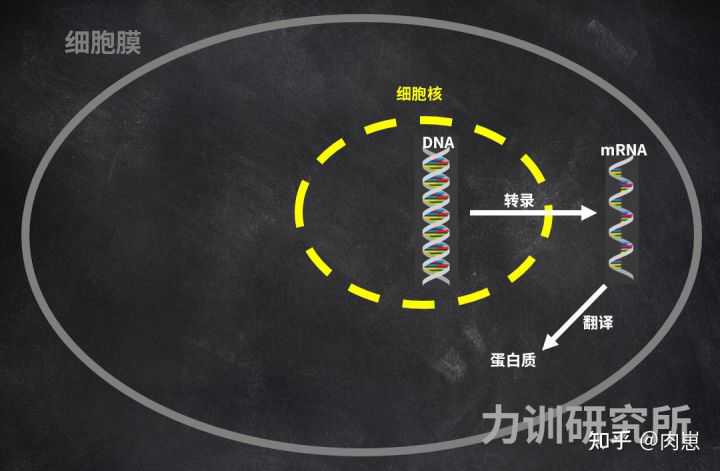
图17:DNA的表达
每种蛋白质都有对应的DNA。
如果我们把蛋白质视为产品,那么DNA就是设计图,蛋白质是依据DNA造出来的[77][78][79][80][81][82][83]。比如我们运输氧气的血红蛋白[84][85]就是生物利用DNA编码出来的[86],人体内无穷多种的蛋白质、酶、身体结构,都是如此。
我们的每个细胞不断凋亡,新的细胞不断产生,这个产生过程,都是DNA表达的结果。我们也可以说,新陈代谢是依靠DNA来进行的,DNA是生命活动的中心。
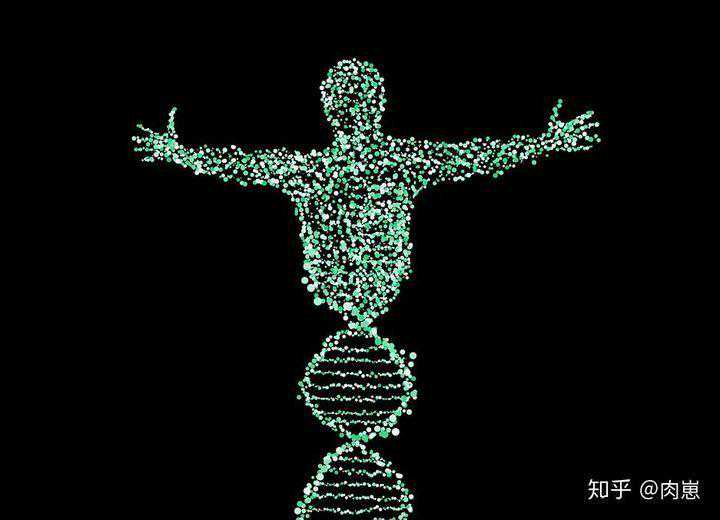
图18:DNA——活动的中心
四、训练:刺激DNA表达我们在前面说了,我们的肌肉中的蛋白质(肌动蛋白/肌球蛋白等)是大量氨基酸以特定方式排列组合而成的。氨基酸的排列组合,依靠mRNA;mRNA是DNA复制的产物。
所以,蛋白质的“制造”过程,最主要是两部:DNA转录为mRNA,mRNA翻译为蛋白质。
从DNA到蛋白质,这也叫基因的表达。
那为什么DNA会开始转录?答案是,有什么东西刺激了它。比如训练,一种施加在肌纤维上的机械外力,也叫机械张力。张,顾名思义,把肌纤维往两边张开、拉开、扯开。
例如在哑铃弯举中,重力作用于哑铃,哑铃把肌纤维往下“扯”,我们自己的骨骼支撑,把肌纤维往上拉,则构成了一个往两边张开的力。

图19:机械张力
机械张力为什么能刺激DNA表达[87][88][89](转录)呢?因为我们有大量的生物感受器,能把外力信号,转变为细胞内的生物信号,这些信号一直传递到DNA上,刺激了DNA的转录,于是我们得到了肌细胞内的蛋白质。
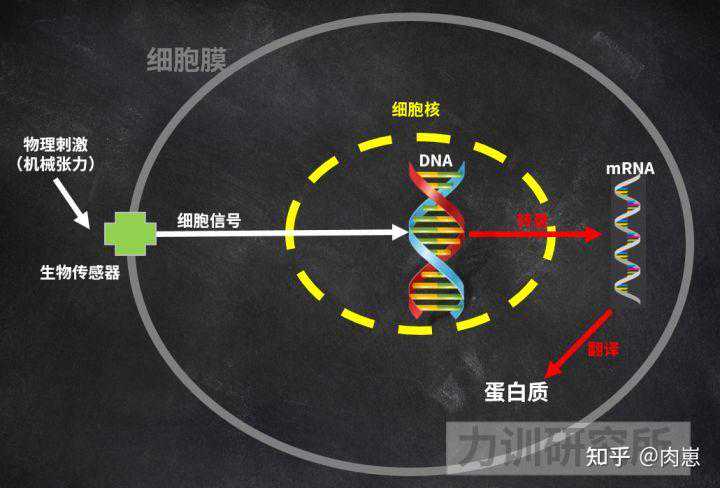
图20:机械刺激与细胞信号
肌肉上能感知机械张力的生物传感器有哪些呢?
例如肋节[90][91][92],它将肌细胞膜与肌原纤维、细胞外基质连接起来,加强肌细胞膜的稳定性和强度,还能感受、侦测到施加于及细胞的外力(例如我们所说的机械张力),将其传导到肌细胞内部,转化为生物信号[16,17];
71整合素[93][94]也是一种横跨细胞膜的受体,它一方面提供连接作用[95],一方面将机械信号从细胞外传递到细胞内。还有磷脂酸(PA)、FAK—粘着斑激酶等也参与机械张力转化为细胞信号的传导传导,就不多说了。
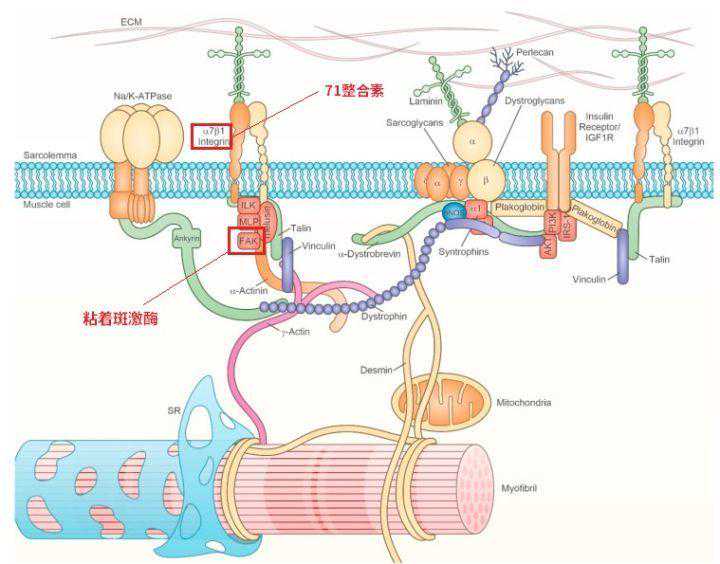
图21:生物传感器
五、训练是如何刺激DNA表达的?答案是细胞信号生物感受器将细胞信号传递到细胞内,引发一系列细胞信号事件。其中最著名、最核心的细胞信号事件,也被称为PI3K/Akt/mTOR路径[96][97][98][99][100]。还有一些别的相对次要的路径(如ERK),碍于篇幅,我们就不在这里说了。
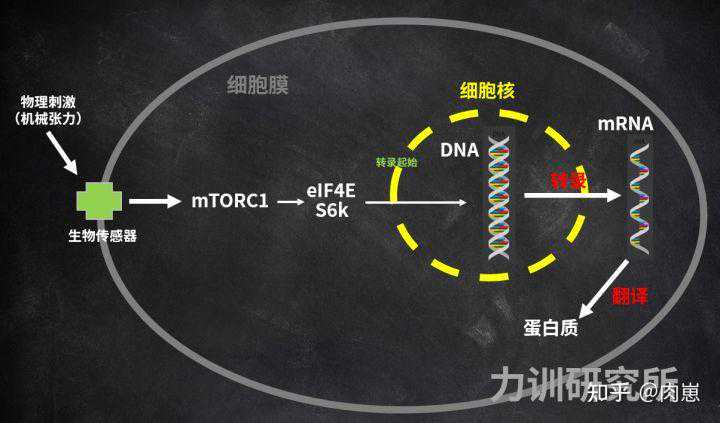
图22:增肌的核心—mTOR路径
mTOR是我们细胞内一种由2549个氨基酸组成的大型信号蛋白[101]——它既是蛋白质,也发挥信号作用。
mTOR全称“哺乳动物雷帕霉素靶蛋白”,它是哺乳动物调节细胞生长、代谢、蛋白质合成等关键生理过程中的重要蛋白[102]。mTOR实际上以mTORC1(复合物1)和mTORC2(复合物2)的形式在人体内存在[103]。
图23:mTORC1
MTORC1主要通过S6K1和eIF4E的磷酸化,来引发DNA表达,促进蛋白质合成[104]。此外,S6k1也提高mRNA的翻译效率[105]。
对于健身者来说,最典型的激活mTORC1的因素,当然是训练。
训练刺激(机械张力)可激活mTORC1,使其磷酸化[106][107][108][109][110][111];在mTOR的下游,S6激酶[112][113][114]和eIF4E(真核生物起始因子4E)[115][116]随之也被磷酸化(红色方框)。
图24:机械张力传递到DNA,引发DNA表达
磷酸化是自然界一种非常普遍的、对蛋白质进行化学修饰的过程[117]。
蛋白质磷酸化有效地增加了其复杂性,远远超过了基因组所赋予的多样性[118]。
在磷酸激酶的作用下,生物将磷酸基团加在蛋白质或蛋白类中间产物上,从而将蛋白质磷酸化(或者去磷酸化)。经化学修饰后的蛋白质,功能/生物活性会显著不同。
目前已知的磷酸激酶多大500多种,可针对超过20000种蛋白质上的25000个点位进行磷酸化[119][120][121][122];磷酸化决定了在正常/病理状态下生物体对刺激的反应[123]。
S6k1是DNA转录因子[124],名至实归。S6k1被激活后,接下来核糖体蛋白6被磷酸化,从而增加了核糖体蛋白与5‘端寡核苷酸(5’-top)mRNA的亲和力,引起了DNA转录[125][126][127],增加蛋白质合成[128][129][130][131]。
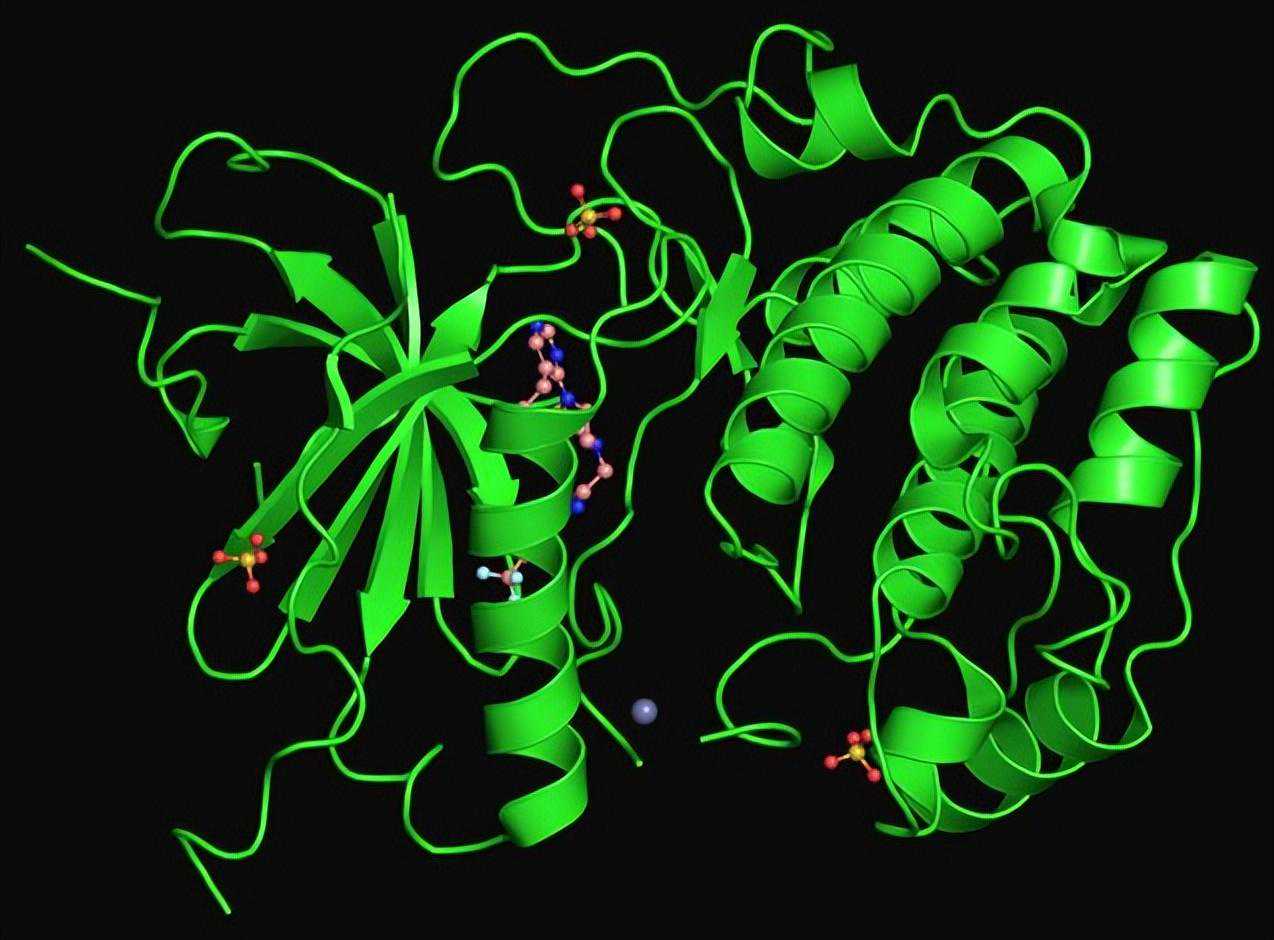
图25:S6k1
反过来,如果蛋白质摄入不足,氨基酸/原料不够,则可以导致核糖体蛋白6的“去磷酸化”[132]。已知的诱导蛋白质去磷酸化的酶超过150种[119]。
研究发现,S6k它的磷酸化水平与增肌之间,存在极强的正相关性,r=0.998。下图纵轴是肌肉增加的幅度,横轴是s6k磷酸化水平,用俗话来说,它们几乎成正比。
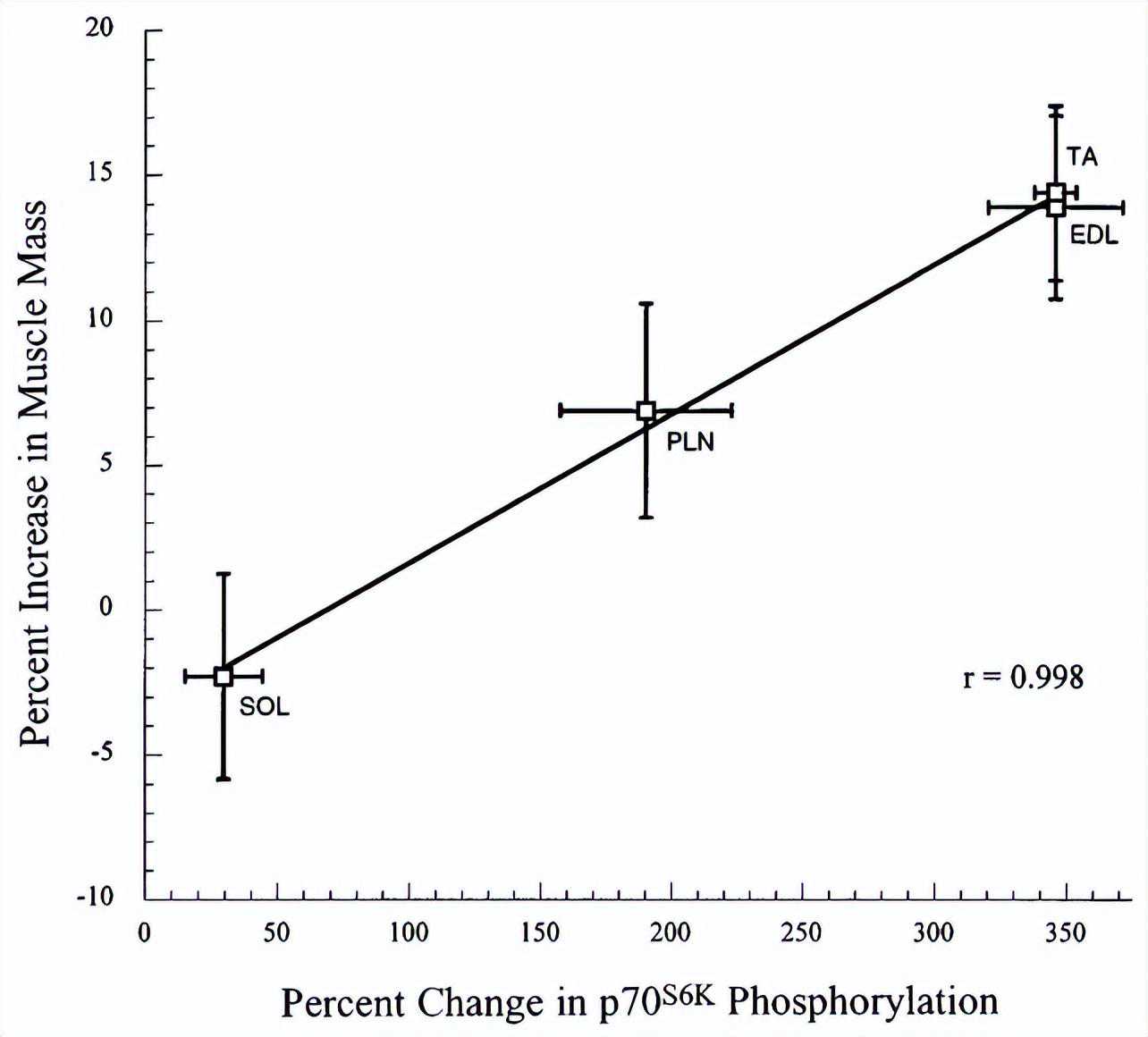
图26:S6k磷酸化与增肌
六、能激活mTOR路径的,不止有训练市面上有一种典型的错误观点,认为训练才能增肌,比如下面图上的这种:

图27:错误观点
注意,图中有2个错误:
错误1:认为增肌的原理是损伤修复。这个错误我们在前面已经解析过了,增肌的主要原理是DNA表达而不是损伤。民间认为损伤增肌,主要是因为损伤是来自于训练,训练能激发DNA表达。
错误2:认为训练是增肌的前提。这也是错的,除了训练,营养[133][134][135][136][137][138][139][140][141]和激素[142][143][144][145][146]也都能激发DNA表达,因为他们的分子路径是高度类似的,原理也相同:都是是通过刺激DNA的表达,来得到更多的肌蛋白。
当然,它们的效果程度不同,这或许是因为三者导致的mTOR磷酸化程度不一样。

图28
我们肌肉中的蛋白质就是这么来的:从激活细胞膜上的受体开始,一个个蛋白/酶依次被激活,最后激活DNA转录,然后mRNA翻译为蛋白质。
许多人很难接受“只打药不练就可以长肌肉”,如果他们知道“只吃蛋白粉不训练也能长肌肉”,估计就更无法接受了。但这是客观事实,不以他们的主观意志为转移。
Liu等人在《临床内分泌与代谢杂志》上发表的了一项以10名健康年轻人为对象的研究,对他们注射氨基酸,观察他们肌细胞内的变化(图30),灰柱“AA”是注射氨基酸后,白色是注射前。纵轴是磷酸化水平。

图30
氨基酸注射,导致p70S6K和eIF4E磷酸化,进而增加了核糖体蛋白S6的磷酸化[147]。S6被磷酸化后,促进了一些在蛋白质合成中起重要作用的核糖体蛋白的合成[148][149]。
这些,也就证明了氨基酸注射或摄入能磷酸化4E-BP1、eIF4E、S6,进而引发蛋白合成:换句话说,单纯的吃蛋白质不运动,也会多多少少长一些肌肉。
除了饮食/营养/训练三者,我们还列举了第四种刺激影响转录因子磷酸化的要素:光。
证据表明,光通过刺激SCN(视交叉上核),来影响ERK、或mTOR路径的下游因子S6核糖激酶的磷酸化[152]。具体的,在我们之前的文章中有过叙述。
参考1.^FrauenfelderH;SligarSG;,254,1598–1603.
2.^FarhathS,HeZ,NakhlaT,,amarkerofgastriccontents,isincreasedintrachea;121:253–259.
3.^StovoldR,ForrestIA,CorrisPA,,abiomarkerofgastricaspirationinlungallografts:;175:1298–1303.
4.^CrapkoM,KerschnerJE,SyringM,‐;117:1419–1423.
5.^KnightJ,LivelyMO,JohnstonN,DettmarPW,;115:1473–1478.
6.^,SchumbrutzkiC.,WortelkampS.,SickmanA.,;75:1454–1462.
7.^,,;3:608–614.
8.^;3:249–275.
9.^RodriguezJ.,GuptaN.,,?;7:300–305.
10.^(RCollPathol)1971;5:29–40.
11.^;55(4):537–608.
12.^MathewsDM,;71(1):151–161.
13.^:;50(11):2266–2275.
14.^;56(5):903–913.
15.^AdibiSA,;52(5):837–845.
16.^;15(6):494–501.
17.^AdibiSA,;60(5):1008–1016.
18.^KaniaRK,SantiagoNA,;252(14):4929–4934.
19.^RaymentI,HoldenHM,WhittakerM,YohnCB,LorenzM,HolmesKC,MilliganRA(1993)Structur:58–65.
20.^RaymentI,RypniewskiWR,Schmidt-B?seK,SmithR,TomchickDR,BenningMM,WinkelmannDA,WesenbergG,HoldenHM(1993)Three-dimensionalstructureofmyosinsubfragment-1::50–58.
21.^RDabrowska,;19(3):343-59.
22.^:Physiological1973;19(3):343-59.
23.^ThorsonJ,;9(3):360–390.
24.^WakabayashiK,SugimotoY,TanakaH,UenoY,TakezawaY,;67(6):2422–2435.
25.^RDBremel,;238(82):97-101.
26.^RicardaHaeger,FelipedeSouzaLeite,;287(1937):20202133.
27.^:frommoleculestosarcomeresto;313(2):
28.^KiisaNishikawa1,SamratDutta2,MichaelDuVall23,BrentNelson4,MatthewJGage5,:;41(1):125-139.Epub2019Jul9.
29.^BBrenner,,mechanical,;82Suppl2:3-16.
30.^MaruyamaK,EbashiS.α-Actinin,;58:13–19.
31.^HondaK,YamadaT,oR,InoY,GotohM,TsudaH,YamadaY,ChibaH,,anovela;140:1383–1393.
32.^Almenar-QueraltA,LeeA,ConleyCA,RibasdePouplanaL,,skeletaltropomodulin,:28466–28475,1999
33.^BroschatKO,WeberA,:8501–8506,1989
34.^FowlerVM,SussmannMA,MillerPG,FlucherBE,(pointed):411–420,1993
35.^GokhinDS,LewisRA,McKeownCR,NowakRB,KimNE,LittlefieldRS,LieberRL,:95–109,2010
36.^LauKF,;87(2):638–642
37.^TsaiCJ;MaB;ShamYY;KumarS;,44,418–427.
38.^TzengSR;KalodimosCGProteindynamicsandallostery:,21,62–67.
39.^ZhuravlevPI;PapoianGAProteinfunctionallandscapes,dynamics,allostery:,43,295–332.
40.^ChanHS,;87(16):6388–6392.
41.^WoodwardCK,;246(13):4114–4121.
42.^ShortleD,ChanHS,;1(2):201–215
43.^VenyaminovSY;RajnavolgyiE;MedgyesiGA;GergelyJ;ZavodszkyPTheroleofinterchaindisulphidebri,67,81–86.
44.^QinH;LimL;SongJProteindynamicsatEphreceptor-ligandinterfacesasrevealedbycrystallography,,5,2.
45.^KarplusM;,260,404–406.
46.^McCammonJA;GelinBR;,267,585–590.
47.^KarplusMTheLevinthalparadox:,2,S69–75.
48.^;29(31):7133–7155.
49.^DillKA,BrombergS,YueK,FiebigKM,YeeDP,ThomasPD,;4(4):561–602.
50.^AckersGK,DoyleML,MyersD,;255(5040):54–63.
51.^AlonsoDO,;30(24):5974–5985.
52.^ShiL,PallerosDR,,1'-bis(4-anilino-5-naphthalenesulfonicacid):;33(24):7536–7546.
53.^Almenar-QueraltA,LeeA,ConleyCA,RibasdePouplanaL,,skeletaltropo-modulin,;274:28466–28475.
54.^CoxPR,,expressionanalysis,andm;63:97–107.
55.^,000;262:12792–12800.
56.^WeberA,PenniseCR,BabcockGG,;127:1627–1635.
57.^,HaberE.,SelaM.,;47:1309–1314.
58.^;181:223–230.
59.^,KommerA.,,;15:242–247.
60.^Neurath,:268‐271.
61.^Hirschberg,,:63‐89.
62.^Suttie,–:459‐477.
63.^Kaufman,,Murtha,P.,Ingolia,,Yeung,,andKellems,
64.^Rothman,:55‐63.
65.^Rothman,,:409‐415.
66.^MathewsM,;17(2):328–338.
67.^SmithAE,MarckerKA,;225(5228):184–187.
68.^LeaderDP,Klein-BremhaarH,;46(1):215–224.
69.^AvivH,BoimeI,:;68(9):2303–2307.
70.^GIACOMONID,;138(3547):1328–1331.
71.^BELOZERSKYAN,;182(4628):111–112.
72.^MarmurJ,:;46(4):453–461.
73.^YcasM,;46(6):804–811.
74.^AvivH,BoimeI,:;68(9):2303–2307.
75.^BoimeI,AvivH,;45(3):788–795.
76.^HeywoodSM,;43(3):470–475.
77.^;40:409–448.
78.^:;13(2):521–531.
79.^McDowellMJ,;45(3):724–733.
80.^;117(3031):89-94.
81.^BenzEJJr,,01Dec1971,50(12):2755-2760
82.^StephenWelle1,KirtiBhatt,;290(3):E409-15.
83.^NeilKubica1,DouglasRBolster,PeterAFarrell,ScotRKimball,ofeukaryoticinitiationf;280(9):7570-80.
84.^DreyerHC,FugitaS,CadenasJG,ChinkesDL,VolpiE,RasmussenB(2006)ResistanceexerciseincreasesAMPKactivityandreduc:613–624
85.^BaarK,EsserK(1999)Phosphorylationofp70S6kcorre:C120–C127
86.^LuisaGorza;MatteoSorge;LauraSeclì;:,10(1),61.
87.^ErvastiJM,OhlieckK,KahlSD,GaverMG,;345:315–9.
88.^ClarkKA,McElhinnyAS,BeckerleMC,:;18:637–706.
89.^DiMauroD,GaetaR,ArcoA,MiliardiD,LentiniS,RunciM,;47(4):605–8.
90.^VattaM,SinagraG,BrunelliL,omyopathies:(Hagerstown)2009;10(2):149–56.
91.^BerthierC,:413–434,1997.
92.^DreyerHC,FugitaS,CadenasJG,ChinkesDL,VolpiE,RasmussenB(2006)ResistanceexerciseincreasesAMPKactivityandreduc:613–624
93.^KubicaN,BolsterDR,FarrellPA,KimballSR,JeVersonLS(2005)Resistanceexerciseincreasesmuscleproteinsynthesisandtranslationofeukaryoticinit:7570–7580.
94.^AthertonPJ,BabrajJ,SmithK,SinghJ,RennieMJ,WackerhageH(2005)SelectiveactivationofAMPK-PGC-1alphaorPKBTSC2-mTORsignalingcanexplainspeciWcadaptiveresponses:786–788
95.^NaderGA(2005)Moleculardeterminantsofskeletalmusclemass::1985–1996
96.^TidballJG(2005):1900–1908
97.^SabersCJ,MartinMM,;270(2):815–22.
98.^SaxtonRA,,Metabolism,;168:960–976.
99.^KimD-H,SarbassovDD,;110(2):163–75.
100.^HolzMK,BallifBA,GygiSP,;123:569–580.
101.^MaXM,YoonSO,RichardsonCJ,JulichK,/;133:303–313.
102.^BodineSC,StittTN,GonzalezM,KlineWO,StoverGL,BauerleinR,ZlotchenkoE,ScrimgeourA,LawrenceJC,GlassDJ,/mTORpathwayisacrucialregul:1014–1019,2001.
103.^GoodmanCA,MiuMH,FreyJW,MabreyDM,LincolnHC,GeY,ChenJ,/proteinkinaseB-indepentactivationofmammaliantargetof:3258–3268,2010.
104.^HornbergerTA,McLoughlinTJ,LeszczynskiJK,ArmstrongDD,JamesonRR,BowenPE,HwangES,HouH,MoustafaME,CarlsonBA,HatfieldDL,DiamondAM,:3091–3097,2003.
105.^HornbergerTA,StuppardR,ConleyKE,FedeleMJ,FiorottoML,ChinER,inositide3-kinase-,:795–804,2004.
106.^KubicaN,BolsterDR,FarrellPA,KimballSR,ofeukaryoticinitiationf:7570–7580,2005.
107.^MiyazakiM,McCarthyJJ,FedeleMJ,isindepentofphosphoinositide3-kinase/:1831–1846,2011.
108.^Pallafacchina,G.,Calabria,E.,Serrano,,Kalhovde,,Schiaffino,S.(2002).AproteinkinaseB-depentandrapamycin-sensitive,25.
109.^Inoki,K.,Li,Y.,Zhu,T.,Wu,J.,Guan,(2002).T,4,648–657.
110.^Hannan,,Thomas,G.,Pearson,(2003).ActivationofS6K1(p70ribosomalproteinS6kinase1)requiresaninitialcalcium-depentprimi,370,469–477.
111.^KimballSR,JeffersonLS,FaddenP,HaysteadTAJ,LawrenceJC1996Insulinanddiabetescauserecipr:C705–C709.
112.^PauseA,BelshamG,GingrasAC,DonzeO,LinTA,LawrenceJC,SonenbergN1994Insulin-depentstimulatio:762–767.
113.^CSRubin,;44:831-87.
114.^abAlonsoA,SasinJ,BottiniN,FriedbergI,FriedbergI,OstermanA,GodzikA,HunterT,DixonJ,andMustelinT(2004).,699–711.
115.^HornbeckPV,KornhauserJM,TkachevS,ZhangB,SkrzypekE,MurrayB,LathamV,andSullivanM(2012).PhosphoSitePlus:acomprehensiveresourceforinvestigatingthestructureandfunctionofexper,D261–270.
116.^KhouryGA,BalibanRC,andFloudasCA(2011).Proteome-widepost-translationalmodificationstatistics:
117.^ManningG,WhyteDB,MartinezR,HunterT,andSudarsanamS(2002).,1912–1934.
118.^PawsonT,andScottJD(2005).,286–290.
119.^SatoshiFujita,HansCDreyer,MicahJDrummond,ErinLGlynn,JersonGCadenas,FumiakiYoshizawa,ElenaVolpi,;582(Pt2):813-23.
120.^JefferiesHBJ,ReinhardC,KozmaSC,'polypyrimidinetract';91:4441–4445.
121.^JefferiesHBJ,ThomasG,α;269:4367–4372.
122.^ThomasG,onseinSwissmouse3T3cells:;103:2137–2144.
123.^GrunerS.,PeterD.,WeberR.,WohlboldL.,,WeichenriederO.,ValkovE.,IgrejaC.,;64:467–479.
124.^Kubica,N.,Bolster,,Farrell,,Kimball,,Jefferson,(2005).Resistanceexerciseincreasesmuscleproteinsynthesisandtranslationofeukaryoticinitiationfactor2B{epsilon},280,7570–7580.
125.^ProudCG,Den:329–341
126.^KleijnM,ScheperGC,VoormaHO,ThomasAA1998:531–544.
127.^ThomasG,onseinSwissmouse3T3cells:;103:2137–2144.
128.^KarlssonHK,NilssonPA,NilssonJ,ChibalinAV,ZierathJR,BlomstrandE(2004)Branched-chainaminoacidsincreasep70:E1–E7
129.^DouglasPaddon-Jones1,MelindaSheffield-Moore,Xiao-JunZhang,ElenaVolpi,StevenEWolf,AsleAarsland,ArnyAFerrando,;286(3):E321-8.
130.^KSmith,JMBarua,PWWatt,CMScrimgeour,[1-13C]leucinestimulateshumanmuscleproteinincorporationofcontinuouslyinfusedL-[1-13C];262(3Pt1):E372-6.
131.^VolpiE,KobayashiH,Sheffield-MooreM,MittorferB,;78:250–8.
132.^PJGarlick,;254(2):579-84.
133.^KoopmanR,CrombachN,GijsenAP,;90:106Y15.
134.^JefferyEscobar,JasonWFrank,AgusSuryawan,HanhVNguyen,ScotRKimball,LeonardSJefferson,;290(4):E612-21.
135.^JCAnthony,TGAnthony,SRKimball,TCVary,s;130(2):139-45.





